R: começar
«R» designa uma linguagem de computador para estatística e com outros usos como qualquer linguagem de programação.
☞ «RStudio» designa uma «app» para Windows, Linux ou Mac, que junta ao R muitas ferramentas como editores, visualizadores, etc; o RStudio é um IDE (Integrated development environment).
instalar em windows
As etapas são:
Instalar o R (biblioteca gigante, em código aberto, da funções estatísticas)
Instalar o RStudio (apropriado para produzir relatórios com R; usamos a versão gratuita)
Pode ser necessário, instalar o executável tirado da net, em modo administrador
Verificação
Executar o RStudio no seu computador.
Copiar o seguinte código R para a janela onde diz «console»; deve surgir um gráfico e uma tabela:
dados = read.csv("https://sweet.ua.pt/pedrocruz/dados/rolhas.csv")
str(dados)
#Construir caixas de bigodes comparativas
boxplot(N_A ~ CLASS, data=dados, pch=20, main="Caixas de bigodes comparativas", xlab=paste("Qualidade da rolha"), ylab=paste("n. total de defeitos"), col="lightgray")
Se a verificação falhar, ainda pode usar o R nesta página, até se resolver o problema.
Disfrute agora da capacidade de rapidamente criar relatórios de tratamento de dados com o RMarkdown:
RMarkdown
A expressão «RMarkdown» designa a combinação de duas tecnologias:
código «R» para cálculos estatísticos e gráficos;
código «Markdown» que serve para criar relatórios rápidos.
Etapas para testar:
Criar uma pasta «bioprojetos» no seu computador.
Abrir o RStudio
menu Session => Set Working Directory => Choose Directory
procurar e escolher a pasta bioprojetos
- Em alternativa, pode mudar a opção em
menu Tools => Global Options => Default Working Directory
começar um projeto novo
Abra o RStudio (se calhar está aberto)
Abra um novo RMarkdown: File => New File => R Markdown
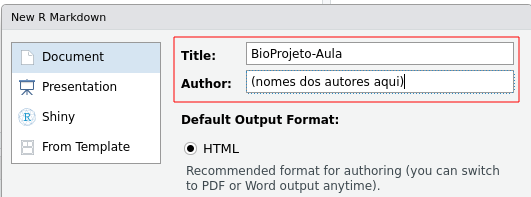
Preencher os dados Title com algo apropriado, e o seu nome:
introduzir dados novos
Para introduzir dados novos, abra um novo ficheiro de dados:
> File => New File => R Markdown (2º rectângulo)
e grave o ficheiro como csv (ex.: robalos.csv, eleicao.csv, prokaryota.csv) na sua pasta «bioprojetos».
Importante
Recomenda-se que os nomes das colunas se devem evitar espaços » » e acentos como «áéí» (é possível mas complica um pouco). Usar o «.» para decimal e «,» para separar colunas também ajuda.
Veja estes exemplos:
tentaculos |
comprimento |
nr_ovos |
area |
ano2001 |
|---|---|---|---|---|
45 |
72.45 |
90 |
Aveiro |
-13.2 |
40 |
81.12 |
84 |
Aveiro |
-3.2 |
23 |
55.20 |
70 |
Águeda |
3.2 |
32 |
48.19 |
64 |
Águeda |
-1.24 |
em que o ficheiro csv tem este conteúdo:
"tentaculos", "comprimento", "nr_ovos", "area", "ano2001"
45, 72.45, 90, "Aveiro", -13.2
40, 81.12, 84, "Aveiro", -3.2
23, 55.2, 70, "Águeda", 3.2
32, 48.19, 64, "Águeda", -1.24
Se usar o Excel deve exportar ou gravar o ficheiro com «csv». Pode ser mais fácil de manusear os dados diretamente nesta ferramenta.
R: tutorial
lista
> x = c(1.2, 1.7, 2.1, 2.2, 2.3)
> x
[1] 1.2 1.7 2.1 2.2 2.3
>
data.frame
Traduzindo à letra: moldura de dados. Trata-se de uma coleção de colunas (tal como uma folha de cálculo) tendo as colunas nomes.
O seguinte comando produz um data.frame com uma coluna chamada comprimento e outra a idade. Estas colunas têm valores indicados por vetores c(…).
> dados = data.frame(comprimento=c(1.2, 1.7, 2.1, 2.2, 2.3), idade=c(10,20,10,12,13))
> dados$comprimento
[1] 1.2 1.7 2.1 2.2 2.3
> dados$idade
[1] 10 20 10 12 13
Para aceder aos valores de uma coluna usa-se a notação dados$comprimento ou, para o caso da idade, dados$idade.
projeto de ensaio
(em desenvolvimento uma nova versão deste material)
Pretende-se introduzir o R + RSTUDIO realizando um projeto de ensaio aguçando a curiosidade do que estará por trás de listas de números ou palavras.
Importante
Pode usar o computador pessoal (ver instalar em windows) ou os computadores da 11.2.7 ou 11.2.8 já com o R e RStudio.
Em aula, efetue as demais etapas abaixo finalizando com a introdução online de resultados.
Posteriormente, e já em TPC, consulte a pasta no elearning para obter cada um dos 4 projetos.
Consulte, quando apropriado, as secções RMarkdown, começar um projeto novo e introduzir dados novos para iniciar um novo projeto.
Este projeto é inspirado no Ex. 1.11, do caderno de exercícios, sendo essa uma ideia a manter: que os projetos são representativos da matéria da UC.
Criar uma pasta «bioprojetos» no seu computador
Abrir o RStudio e a indicar a pasta de trabalho com as etapas:
menu Session => Set Working Directory => Choose Directory
procurar e escolher a pasta bioprojetos
Transfira o ficheiro salinidade.csv para a sua pasta «bioprojetos».
Transfira o ficheiro BioProjeto-aula-v2-latin1.Rmd para a sua pasta «bioprojetos».
No RStudio, abra esse ficheiro: File => Open File
Siga, agora, as instruções nele contidas.